R 패키지중에 DiagrammeR라는 다이어그램(diagram)을 그릴 수 있게 해주는 것이 있습니다. 다이어그램은 플로우차트(flow chart), 간트 차트(gantt chart), 시퀀스 다이어그램 (sequence diagram)같은 것입니다.

다이어그램을 그릴 때 쓰는 도구는 Visio (Windows 쓰시는 분들) 아니면 OmniGraffle (Mac 쓰시는 분들) 아니면 PowerPoint 와 같이 손으로 그리는 것들이 있고 GraphViz 또는 Mermaid와 같이 정해진 문법을 텍스트로 입력하면 해석해서 시각적으로 표현해주는 도구가 있습니다.
DiagrammeR는 GraphViz와 Mermaid를 묶어서 연동해 놓은 패키지인데 3가지 방식으로 그래프를 그리게 해줍니다.
- Mermaid 문법을 텍스트로 입력한 후 텍스트를 해석해서 렌더링
- GraphViz 문법을 텍스트로 입력한 후 텍스트를 해석해서 렌더링
- R함수를 사용해서 노드와 엣지를 구성하고 렌더링
패키지를 사용하던 중 GraphViz는 원래 C로 만들어진 binary이므로 R에서 연동해서 사용할 수 있습니다만 Mermaid는 Javascript로 만들어져서 이 두가지를 어떻게 한꺼번에 연동해서 그래프를 시각적으로 표현하게 했는지 갑자기 궁금했습니다.
그래서 살펴봤더니 GraphViz.js와 Mermaid.js를 가져다 연동한 것이고 렌더링된 결과를 표현할 때는 htmlwidgets 패키지를 사용하는 것입니다.
그러니까 결국 웹브라우저를 열고 Javascript를 이용해서 렌더링하는 방식입니다.
그래서 R-GUI에서 렌더링을 하게 되면 웹브라우저가 열립니다. RStudio에서 렌더링을 시도하면 연동이 되어서 웹브라우저가 실행되지 않고 그래프 패널에 바로 렌더링된 결과를 표현해 줍니다. 위에 넣어 놓은 스크린샷 이미지에서 확인할 수 있습니다.
앞서 말한 렌더링을 하는 세가지 방식중에 Mermaid 문법을 사용하는 것과 GraphViz 문법을 사용하는 것은 RStudio에 연동되어서 파일을 편집할 수 있게 제공하고 있기때문에 R 코드에서 DiamgrammeR 패키지를 로딩할 일이 없게 만들기도 합니다.
RStudio는 DiagrammeR를 연동해서 .mmd 확장자를 가지는 Mermaid 파일과 .dot 확장자를 가지는 Graphviz dot 파일을 바로 편집하고 코드 하일라이트도 되고 렌더링할 수 있도록 해줍니다.
아래의 링크에서 내용을 확인할 수 있습니다.
https://blog.rstudio.com/2015/05/01/rstudio-v0-99-preview-graphviz-and-diagrammer/
위의 3가지 방식 중 2가지는 앞서 말씀드린 것 처럼 RStudio와 연동으로 인해 DiagrammeR 패키지의 함수를 사용할 일이 없게 만들지만 DiagrammeR 함수를 이용한 방식의 렌더링을 사용하면 data.frame에 있는 데이터를 연동해서 그래프를 그릴 수 있습니다. 이게 가장 큰 장점이지요.
아래는 각각의 방법으로 만든 간단한 장난감 예제입니다.
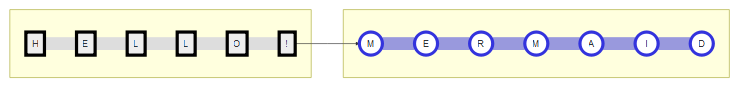
Mermaid 문법을 이용한 렌더링
Mermaid 문법을 이용한 예제입니다.
library(DiagrammeR)
DiagrammeR('
graph LR
subgraph ""
N01[H] --- N02
N02[E] --- N03
N03[L] --- N04
N04[L] --- N05
N05[O] --- N06
N06[!]
end
N06 --> N07
subgraph ""
N07((M)) --- N08
N08((E)) --- N09
N09((R)) --- N10
N10((M)) --- N11
N11((A)) --- N12
N12((I)) --- N13
N13((D))
end
classDef box1 color:white,fill:#eee,stroke:#000,stroke-width:5px
classDef box2 color:white,fill:#fff,stroke:#33d,stroke-width:5px
class N01,N02,N03,N04,N05,N06 box1
class N07,N08,N09,N10,N11,N12,N13 box2
linkStyle 0 stroke:#ddd,stroke-width:20px
linkStyle 1 stroke:#ddd,stroke-width:20px
linkStyle 2 stroke:#ddd,stroke-width:20px
linkStyle 3 stroke:#ddd,stroke-width:20px
linkStyle 4 stroke:#ddd,stroke-width:20px
linkStyle 6 stroke:#99d,stroke-width:20px
linkStyle 7 stroke:#99d,stroke-width:20px
linkStyle 8 stroke:#99d,stroke-width:20px
linkStyle 9 stroke:#99d,stroke-width:20px
linkStyle 10 stroke:#99d,stroke-width:20px
linkStyle 11 stroke:#99d,stroke-width:20px
')
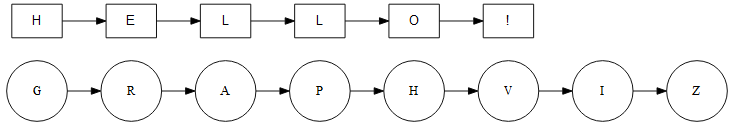
GraphViz 문법을 이용한 렌더링
GraphViz 문법을 이용한 예제입니다.
grViz('
digraph boxes_and_circles {
graph [overlap = true, fontsize = 12]
rankdir="LR"
subgraph cluster_2 {
node [shape = circle,
fixedsize = true,
width = 0.9]
color=none
rank=same
N07[label="G"]
N08[label="R"]
N09[label="A"]
N10[label="P"]
N11[label="H"]
N12[label="V"]
N13[label="I"]
N14[label="Z"]
N07->N08->N09->N10->N11->N12->N13->N14
}
subgraph cluster_1 {
node [shape = box,
fontname = Helvetica]
color=none
rank=same
N01[label="H"]
N02[label="E"]
N03[label="L"]
N04[label="L"]
N05[label="O"]
N06[label="!"]
N01->N02->N03->N04->N05->N06
}
}
')
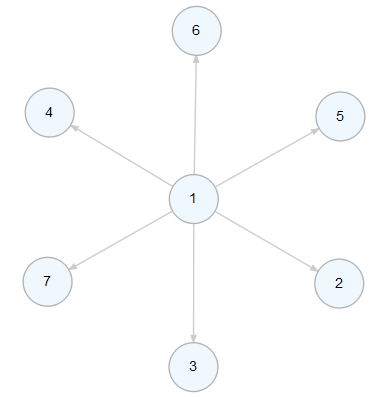
GrammerR의 R 함수를 이용한 렌더링
전통적인 스타일의 R 함수를 사용한 예제입니다. 아래의 함수들은 DiagrammeR 최신 패키지를 설치해야만 됩니다. 최근에 함수 이름에 변화가 있었던 모양입니다.
library(DiagrammeR)
nodes <-
create_node_df(
n = 7,
type = "number")
edges <-
create_edge_df(
from = c(1, 1, 1, 1, 1, 1),
to = c(2, 3, 4, 5, 6, 7),
rel = "related")
graph <-
create_graph(
nodes_df = nodes,
edges_df = edges)
render_graph(graph)
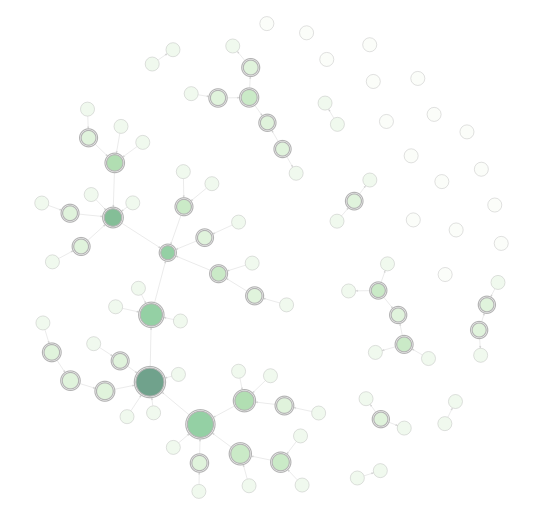
dplyr와 연동한 DiagrammeR
역시 RStudio에서 만은 부분을 기여한 패키지인 만큼 dplyr 스타일의 매우 스타일리쉬한 파이프라인 형태의 코딩도 지원합니다.
library(DiagrammeR)
example_graph <-
create_graph() %>%
add_pa_graph(
n = 50,
m = 1,
set_seed = 23) %>%
add_gnp_graph(
n = 50,
p = 1/100,
set_seed = 23) %>%
join_node_attrs(
df = get_betweenness(.)) %>%
join_node_attrs(
df = get_degree_total(.)) %>%
colorize_node_attrs(
node_attr_from = total_degree,
node_attr_to = fillcolor,
palette = "Greens",
alpha = 90) %>%
rescale_node_attrs(
node_attr_from = betweenness,
to_lower_bound = 0.5,
to_upper_bound = 1.0,
node_attr_to = height) %>%
select_nodes_by_id(
nodes = get_articulation_points(.)) %>%
set_node_attrs_ws(
node_attr = peripheries,
value = 2) %>%
set_node_attrs_ws(
node_attr = penwidth,
value = 3) %>%
clear_selection() %>%
set_node_attr_to_display(
attr = NULL)
#> `select_nodes_by_id()` INFO: created a new selection of 34 nodes
#> `clear_selection()` INFO: cleared an existing selection of 34 nodes
example_graph %>%
render_graph(layout = "nicely")
추가로 네트워크 분석 관련 패키지인 igraph에서 생성한 객체를 DiagrammeR 형태로 변환하는 함수들도 제공합니다. 사용해 보지는 않았습니다. ^^;
